经过多年的延迟FDA批准了Teva的通用EpiPen
2022年3月14日, 国际学术期刊Gut在线发表了复旦大学生命科学学院徐书华教授团队的研究成果“Improved NGS variant calling tool for the pRSS1-pRSS2 locus”。该研究开发了新的遗传变异检测工具NGS.pRSS1-2caller,显著提升了胰蛋白酶原基因座pRSS1-pRSS2在短读长的二代测序(NGS)技术中遗传变异检测的准确度与灵敏度,使其能够应用于临床诊断,并且发现了胰腺炎的易感性可能与该基因座的序列结构密切相关。
胰蛋白酶(trypsin)是一种重要的消化酶,能够帮助人类消化食物中的蛋白质。胰蛋白酶原(trypsinogen)是胰蛋白酶的前体,它在胰脏中合成,作为胰液的成分而分泌。其中,阳离子与阴离子胰蛋白酶占了人类总胰蛋白酶原的90%以上,分别受到人类7号染色体上pRSS1和pRSS2基因的编码。pRSS1与pRSS2基因在胰脏中特异性表达,且pRSS1上的一些位点变异(包括单核苷酸突变与拷贝数变异)会导致胰腺炎的产生。尽管目前第二代测序技术已较为普及,但是研究发现传统的NGS遗传变异检测会造成pRSS1和pRSS2上产生大量的假阳性结果,从而造成遗传诊断的误判。研究团队通过对pRSS1-pRSS2序列结构的解析,发现了假阳性结果的产生是由于人类中存在两种结构类型的单倍型—3基因单倍型与5基因单倍型。这两种单倍型都是由同源的胰蛋白酶原基因重复(gene duplication)产生的,两者的差别在于5基因单倍型比3基因单倍型多了两个假基因(pRSS3p2和TRY7)。而当这两个假基因的NGS测序片段(sequencing read)比对到3基因单倍型的参考基因组上时,就会由于序列的同源性被错误比对到pRSS1上,从而产生大量的假阳性。相反地,如果用5基因单倍型作为参考基因组就能较好地避免假阳性的产生(图1A)。基于此,研究团队开发了新的工具(NGS.pRSS1-2caller)用于pRSS1-pRSS2基因座的遗传变异检测。相比于传统的遗传变异检测软件GATK,评估显示NGS.pRSS1-2caller不仅大幅提升了pRSS1-pRSS2基因座检测的准确度与灵敏度,还能同时实现对序列结构(拷贝数变异)与单核苷酸突变的判读(图1B)。最后,研究团队将NGS.pRSS1-2caller应用在千人基因组数据中,结合已知的胰腺炎风险位点进行连锁分析,发现了在欧亚人群的胰腺炎易感性与pRSS1-pRSS2座的单倍型结构存在紧密联系。
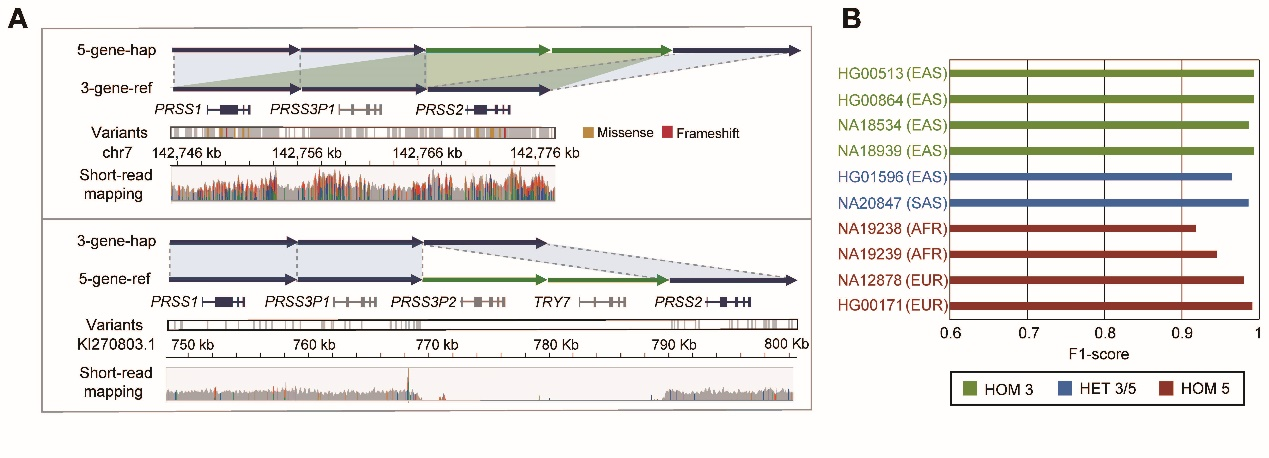
图1.NGS测序片段的比对(A)与NGS.pRSS1-2caller在真实数据中的评测(B)。
该研究工作由复旦大学徐书华团队青年研究员楼海一博士、中国科学院上海营养与健康研究所博士生谢波和王亦民作为共同第一作者主要实施和完成,博士生高扬参与了数据处理分析工作。楼海一博士与徐书华教授为该文共同通讯作者;复旦大学生命科学学院为第一单位与通讯单位;复旦大学附属中山医院与人类表型组研究院为通讯作者单位。
该项工作得到了国家自然科学基金委、人类表型组上海市市级重大专项、中国科学院动物进化与遗传前沿交叉卓越创新中心、英国皇家学会牛顿基金、上海市科委等多项基金的资助。
原文链接:https://gut.bmj.com/content/early/2022/03/13/gutjnl-2022-327203
-
合成微生物使科学家们能够研究古老的进化神秘
2022-04-29 -
加强了恰帕斯高地土着居民的领土管理合作进程
2022-04-29 -
介绍CNVP,IUCN的新成员
2022-04-29 -
销售人员为肥胖客户推荐更多圆形产品
2022-04-29 -
黄石麋鹿的疾病隐藏成本 USU生态学家说 布鲁氏菌病会减少怀孕
2022-04-29 -
研究人员设计“智能”表面以排斥一切 但针对有益的例外
2022-04-29 -
大麻使用的性别差异开始在动物和人类的大脑研究的帮助下得到解释
2022-04-29 -
缅甸开始在全国红色名单上工作
2022-04-29 -
转染市场五年后将达到10亿美元
2015-07-08















