经过多年的延迟FDA批准了Teva的通用EpiPen
2021年8月12日,Nature Communications在线发表了中国农业科学院(深圳)农业基因组研究所(以下简称“基因组所”)左二伟团队与中国科学院脑科学与智能技术卓越创新中心孙怡迪研究组的研究论文,题为“Optimization of C-to-G base editors with sequence context preference predictable by machine learning methods”。该研究优化得到高效率、高保真的新型OpTI-CGBEs,并开发出能够准确预测OpTI-CGBEs编辑效果的深度学习模型CGBE-SMART。
 研究背景
研究背景单碱基编辑技术是近年来发展的一种新型基因编辑技术,它基于CRISpR/Cas9系统,能够在不打断DNA双链的前提下,精准地实现DNA中单个碱基的替换,在生物研究和临床医疗中应用广泛。
然而目前已经开发并得到广泛应用的碱基编辑器只能将C·G碱基对替换为T·A碱基对(C→T),或者将A·T替换为G·C(A→G),即只能修复由C>T或者A>G导致的遗传表型或者疾病,而对于其它类型的单碱基突变束手无策。
2020年,科研人员在胞嘧啶碱基编辑器(CBE)的基础上,研发了能够将胞嘧啶转换为鸟嘌呤的碱基编辑器(C-to-G base editor, CGBE),但CGBE编辑器技术仍处于初步阶段,其特异性、保真性以及编辑特点仍需进一步研究。
研究结果2021年,中国农业科学院深圳农业基因组研究所左二伟研究组与中国科学院脑科学与智能技术卓越创新中心孙怡迪研究组合作开发了高效率、高保真的新型OpTI-CGBEs并建立机器学习模型CGBE-SMART。该研究成果将进一步加速CGBE的应用研究,为科研人员提供更高效、脱靶率低的基因编辑工具。
研究方法
首先通过筛选不同物种来源的UNGs、密码子优化等方式获得OpTI-CGBEs,并在全基因组及转录组范围内研究其保真性。接着通过内源位点及文库水平研究发现OpTI-CGBEs在WCW motif中的C-to-G编辑效率更高,研究人员进一步开发出TCW motif偏好性的eA3A-OpTI-CGBEs,偏好CCN motif的hA3G-OpTI-CGBEs和hA3G-CTD-OpTI-CGBEs等编辑器,或者用可识别NG pAM的Cas9n-NG、spGn和xCas9n替换OpTI-CGBEs中的nCas9,分别构建了不同CGBEs版本,扩大了CGBE的可编辑范围。
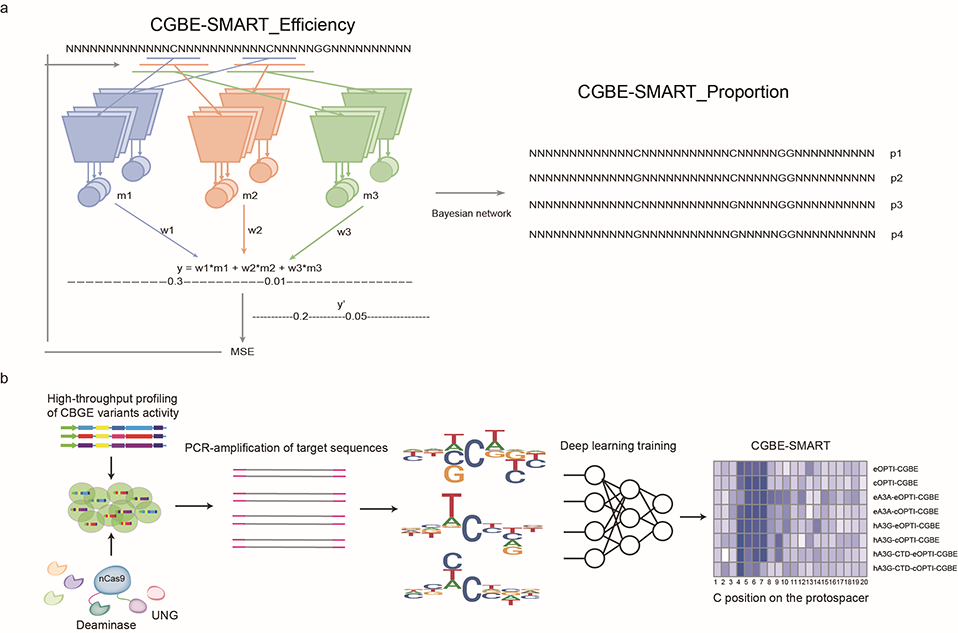
为方便其他研究人员选择合适的C-to-G碱基编辑器和高效预估编辑效率,研究人员建立预测不同C-to-G碱基编辑器编辑效果的深度学习模型CGBE-SMART。它不仅能够准确预测C-to-G编辑效率,而且与之前的预测模型相比,在预测C-to-T编辑效果中也有极为出色的表现。CGBE-SMART结合神经网络和概率图模型,为每一个编辑位置独立训练一套参数来预测该位置上的编辑效率。该模型使用了大小不同的卷积核,建立一组基础单元网络对编辑位置周围的碱基进行特征提取和效率预测。最终研究人员将不同基础单元网络的预测结果用一套习得的参数进行加权平均。模型以编辑位点附近的40bp作为输入,通过神经网络预测出guide RNA结合位置1至20的编辑效率并进一步利用贝叶斯网络预测不同编辑结果的占比。研究人员还利用构建的OpTI-CGBEs在小鼠胚胎中高效对Tyr毛色基因进行编辑。
基因组所助理研究员袁堂龙、博士后闫娜娜、硕士研究生郑基坛、刘敬、科研助理李娜娜、中国科学院脑科学与智能技术卓越创新中心费天一和孟娟为该论文的共同第一作者,基因组所左二伟研究员和中国科学院脑科学与智能技术卓越创新中心孙怡迪研究员为共同通讯作者。该研究得到了基金委,农科院,深圳市的资助。
相关论文信息:https://www.nature.com/articles/s41467-021-25217-y
-
合成微生物使科学家们能够研究古老的进化神秘
2022-04-29 -
加强了恰帕斯高地土着居民的领土管理合作进程
2022-04-29 -
介绍CNVP,IUCN的新成员
2022-04-29 -
销售人员为肥胖客户推荐更多圆形产品
2022-04-29 -
黄石麋鹿的疾病隐藏成本 USU生态学家说 布鲁氏菌病会减少怀孕
2022-04-29 -
研究人员设计“智能”表面以排斥一切 但针对有益的例外
2022-04-29 -
大麻使用的性别差异开始在动物和人类的大脑研究的帮助下得到解释
2022-04-29 -
缅甸开始在全国红色名单上工作
2022-04-29 -
转染市场五年后将达到10亿美元
2015-07-08















